RNA Seqとマイクロアレイの大きな違いは、RNA Seq(RNA Sequencing)が新規RNAやRNAバリアントを解析できるのに対し、マイクロアレイは既知のRNAプローブを用いてトランスクリプトームを解析できる点です。
さらに、RNA Seqはシーケンシングに基づく手法であるのに対し、マイクロアレイはハイブリダイゼーションに基づく手法です。
RNA Seqとマイクロアレイは、生物の全mRNA発現量であるトランスクリプトームの解析に用いられる2つの技術です。
RNA Seqとマイクロアレイを用いることで、特定の細胞ステージで形成されるRNA分子の種類を特定することができる。
RNA Seqとは?
RNA Seq (RNA sequencing) は、サンプル中の RNA 分子の配列を決定する技術です。
RNA Seqでは、cDNA分子のハイスループットなショットガンシーケンスを行います。
cDNAは、RNA分子の逆転写から得られます。
次世代シーケンサーでは、cDNAの塩基配列を決定する。
RNA Seqでは、RNAの配列とその相対的な存在量を決定することができる。
RNA Seqは信頼性の高い手法であり、感度範囲も広い。
そのため、希少なRNA配列や低濃度のRNA配列を検出することができます。
RNA Seqの特徴は、新規RNA配列やスプライシングバリアントの同定が可能なことです。
マイクロアレイとは
マイクロアレイは、特定の生物の遺伝子発現を解析するために広く用いられている技術です。
マイクロアレイでは、サンプル中のRNA分子とハイブリダイズするプローブを使用する。
一本鎖プローブは、チップと呼ばれる固体表面に取り付けられており、そこに蛍光標識されたRNA試料がハイブリダイズされる。
プローブの設計には、ゲノム配列のデータを利用することができる。
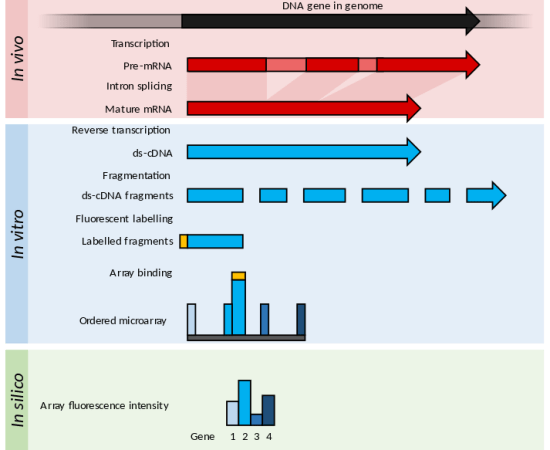 図2:マイクロアレイのプロセス
図2:マイクロアレイのプロセス
マイクロアレイは、遺伝子発現解析において、迅速かつ簡便に実験を行うことができる最適な手法です。
しかし、スプライシングバリアントも検出できるようにプローブを設計する必要がある。
RNA Seqとマイクロアレイの類似性
- RNA Seqとマイクロアレイは、遺伝子発現解析に使用される2つの技術です。
- RNA Seqとマイクロアレイは、遺伝子発現解析に使用される技術であり、ある時点のサンプルに存在するRNA分子の種類を検出することができる。
- RNAの配列に依存する。
- 両手法とも、サンプル中のRNAの一次配列と相対量を決定することができる。
- 両手法とも再現性が高い。
RNA Seqとマイクロアレイの違い
定義
RNA Seqはサンプル中のRNA配列を検出するシーケンシング技術、マイクロアレイはサンプル中の特定のRNA配列の存在を検出するために使用されるハイブリダイゼーション技術であることを意味する。
ベースとなる
RNA Seqはシーケンシングに基づく手法であり、マイクロアレイは既存のプローブを用いたハイブリダイゼーションに基づく手法です。
新規配列の同定
マイクロアレイは既知の配列しか特定できないが、RNA Seqはサンプルに存在する新しいRNA配列を特定することができる。
感度
RNA Seqの感度は高いが、マイクロアレイの感度は比較的低い。
精度
RNA Seqデータの精度は高いが、マイクロアレイデータの精度は比較的低い。
SNP検出
RNA Seqは低濃度RNAのde novo SNPsを除いてSNPを検出できるが、マイクロアレイはSNPを検出できない。
コスト
RNA Seqはバイオインフォマティクス解析が必要なため高価($300-$1000/sample)であるのに対し、マイクロアレイは安価($100-200/sample)です。
結論
RNA Seq は、トランスクリプトームに存在する RNA 配列を決定するために使用される配列ベースの技術であり、マイクロアレイはトランスクリプトーム内の特定の配列の存在を検出するために特定のプローブを使用するものです。
マイクロアレイでは、新規のRNA配列や存在量の少ないRNA配列は検出できない。
しかし、RNA Seqでは、サンプル内のすべてのRNA配列を同定することができます。
したがって、RNA Seqとマイクロアレイの主な違いは、検出の種類にあります。
