サンガーシーケンシングとパイロシーケンシングの大きな違いは、サンガーシーケンシングがジデオキシ鎖終結法を用いたDNAシーケンシング手法であるのに対し、パイロシーケンシングはsequencing by synthesisの原理に基づいたDNAシーケンシング手法である点です。
したがって、サンガーシークエンシングでは、全DNA断片を増幅した後、キャピラリー電気泳動でヌクレオチドの同定を行い、パイロシークエンシングでは、合成時にピロリン酸を遊離させてヌクレオチドの同定を行うというものです。
サンガーシーケンスとパイロシーケンスは、DNA配列決定の2つの方法で、前者はほとんどのターゲットに対する「ゴールドスタンダード」であり、後者は従来のサンガーシーケンス法に代わる最初の方法です。
主な対象分野
- サンガーシークエンスとは
– 定義、プロセス、重要性 - パイロシーケンスとは
– 定義、プロセス、重要性 - サンガーシーケンスとパイロシーケンスの共通点
– 共通点の概要 - サンガーシーケンス・パイロシーケンスの違いについて
– 主な違いの比較
この記事の重要な単語
DNAシーケンス、PCR、ピロリン酸、パイロシーケンス、サンガーシーケンス、高感度
サンガーシークエンスとは
サンガーシークエンシングは、1977年にFredric Sangerによって開発された第一世代のDNA配列決定法です。
また、サンガーシーケンスの基本は、ジデオキシ鎖切断法です。
サンガーシークエンス – 手順
サンガーシークエンスでは、DNAポリメラーゼはin vitroのDNA合成の際に鎖終結ジデオキシヌクレオチド(ddNTP)を選択的に取り込む役割を担っている。
そこで、PCRによりジデオキシヌクレオチド(ddNTPs)をアンプリコンに蛍光標識する。
ここで、ddATPは緑の色素で、ddGTPは黄色の色素で、ddCTPは青の色素で、ddTTPは赤の色素でラベルされている)その後、得られたアンプリコンをキャピラリー電気泳動で分離し、蛍光ラベルされたヌクレオチドを検出する。
図1:サンガーシークエンス法
サンガーシークエンス – 重要性
しかし、サンガーシーケンス法には、長いシーケンスを処理できない、少ないサンプルの並列解析ができない、サンプル調製の完全自動化ができない、コストが高い、シーケンスエラー、感度が低い(10-20%)、低レベル変異アレルの検出には不十分、などの制限があります。
これらの制限にもかかわらず、多くの臨床処置におけるシーケンシングの「ゴールドスタンダード」です。
パイロシークエンスとは
パイロシークエンシングは、従来のサンガーシークエンシングに代わる初めての次世代シーケンサーです。
王立工科大学(KTH)で開発された次世代シーケンシングの一種です。
また、この方法は、プライマー誘導型DNAポリメラーゼ触媒によるヌクレオチドの組み込みの際に放出されるピロリン酸(PPi)をルミノメーターで検出することに基づいている。
パイロシークエンス – 手順
この方法では、取り込まれたヌクレオチドを正確に検出するために、一般に4つの酵素が使用されます。
DNAポリメラーゼ、ATPスルフリラーゼ、ルシフェラーゼ、アピラーゼの4つです。
また、シークエンスプライマーは、一本鎖DNAビオチン標識鋳型にハイブリダイズする。
さらに、4つのデオキシヌクレオチド三リン酸(dNTP)、アデノシン5’ホスホ硫酸(APS)およびルシフェリンが反応混合物中の基質となる。
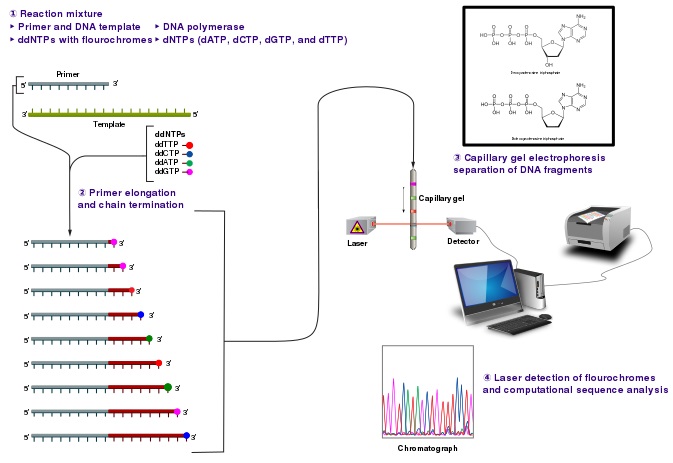 図2:パイロシークエンス法
図2:パイロシークエンス法
重合カスケードが始まると、ポリメラーゼによるヌクレオチドの取り込みの結果、無機PPiが放出される。
しかし、放出されるPPiの量は、各サイクルにおいて取り込まれたヌクレオチドの量と等モルです。
その後、ATPスルフリラーゼは、APSの存在下で、放出されたPPiを定量的にATPに変換する。
生成されたATPは、ルシフェラーゼ酵素を介したルシフェリンのオキシルシフェリンへの変換を駆動する。
また、この反応では、ATPの量に比例して可視光が発生する。
そして、この光は波長560nmで検出することができる。
さらに、アピラーゼ酵素の主な働きは、ATPと反応混合物中の非組み込みdNTPを連続的に分解することである。
したがって、新しいdNTPを65秒という一定の時間間隔で1つずつ反応に加える必要がある。
加えたヌクレオチドがわかっているので、鋳型の配列を決定することができる。
パイロシークエンス -重要性
パイロシークエンシングは、高精度、並列処理、自動化が容易であり、広く応用できる技術です。
また、標識プライマー、標識ヌクレオチド、ゲル電気泳動などを使用する必要がありません。
さらに、確認シーケンシングとde novoシーケンシングの両方に適しています。
さらに、パイロシーケンスの最大の特徴は、その配列深度にあり、バリアントを高感度で検出することが可能です。
しかし、この技術の最大の欠点は、数百塩基までの配列決定にしか適さないことです。
サンガーシークエンスとパイロシークエンスの類似点
- サンガーシークエンシングとパイロシークエンシングは、DNAの塩基配列を決定する2つの方法です。
- サンガーシークエンシングとパイロシークエンシングは、DNAの塩基配列を決定する2つのアプローチです。
- どちらもより小さなDNA断片の塩基配列を決定するのに適しています。
- しかし、そのシーケンス手順や利点により、それぞれ独自の用途があります。
サンガーシークエンスとパイロシークエンスの違い
定義
サンガーシーケンスとは、鎖終結ジデオキシヌクレオチドの選択的導入によるDNA配列決定法、パイロシーケンスとは、合成によるDNA配列決定法です。
シーケンシングの種類
サンガーシーケンスは第一世代のシーケンシング手法であり、パイロシーケンスは第二世代のシーケンシング手法である次世代シーケンシングケミストリーです。
相関関係
さらに、サンガーシーケンスは従来の方法であり、ほとんどのターゲットに対して「ゴールドスタンダード」であるが、パイロシーケンスは従来のシーケンサー法に代わる最初の方法です。
発明
1977年にFrederick SangerらがSanger sequencingを、1996年にPål Nyrénとその学生Mostafa Ronaghiがパイロシーケンスを、ストックホルム王立工科大学で初めて開発した。
商品化
サンガーシーケンスはApplied Biosystems社が最初に商品化したものであるが、パイロシーケンスはRoche社の454やGS FLX Titaniumプラットフォームで使用されている。
原則
サンガーシークエンシングとパイロシークエンシングの大きな違いは、サンガーシークエンシングがジデオキシ鎖終結法を用いるのに対し、パイロシークエンシングはsequencing by synthesisの原則に基づくことである。
ヌクレオチドの同定
サンガーシークエンスでは、DNA断片全体を増幅した後、キャピラリー電気泳動でヌクレオチドの同定を行いますが、パイロシークエンスでは、合成時にピロリン酸を遊離させてヌクレオチドの同定を行います。
検出
さらに、サンガーシークエンスでは蛍光灯の光を、パイロシークエンスでは560nmの可視光線を検出することができる。
DNA断片の長さ
さらに、サンガーシークエンスでは800~1000塩基対、パイロシークエンスでは300~500塩基対まで読み取ることが可能です。
意義
サンガーシークエンスは多くのステップを要する複雑なプロセスであるのに対し、パイロシークエンスはステップ数が少なく、複雑なプロセスではありません。
感度
また、Sanger sequencingとPyrosequencingのもう一つの違いは、Sanger sequencingは感度が低く、Pyrosequencingは感度が高いことである。
結論
サンガーシーケンスは、第一世代のシーケンシングアプローチで、従来のシーケンシング方法です。
また、多くのターゲットに対して「ゴールドスタンダード」です。
しかし、これはジデオキシ鎖終結法に続いてキャピラリー電気泳動を使用する。
一方、パイロシーケンスはサンガーシーケンスに代わる最初の手法であり、次世代シーケンサーの一種です。
さらに、感度が高く、ステップ数が少ないのが特徴です。
一般的には、DNA断片をそのまま合成する際にヌクレオチドを決定するsequencing-by-synthesis方式を採用している。
したがって、サンガーシーケンスとパイロシーケンスの主な違いは、シーケンシングの方法とその利点になります。
